Tutorial: Detección de anomalías cardiacas con Autoencoders en Python
En este tutorial veremos como usar el Deep Learning en el área de la salud, entrenando un Autoencoder capaz de detectar anormalidades cardiacas.
Al final de este tutorial se encuentra el enlace para descargar el código fuente.
Así que ¡listo, comencemos!
Tabla de contenido
Video
Como siempre, en el canal de YouTube se encuentra el video de este post:
Introducción
En un tutorial anterior vimos un cómo realizar la detección de fraudes usando autoencoders y en este caso veremos cómo usar esta misma arquitectura del Deep Learning en una aplicación al área de la salud.
En particular veremos cómo usar los autoencoders para detectar anomalías en señales cardiacas, provenientes de electrocardiogramas (ECGs). Con este autoencoder podremos entonces determinar si el paciente es sano o si tiene algún tipo de enfermedad.
El problema a resolver
El objetivo de este tutorial es entrenar un autoencoder que sea capaz de detectar la presencia o ausencia de irregularidades en el ritmo cardiaco, a partir de señales ECG. En el caso de la presencia de alguna anomalía clasificaremos al sujeto como “anormal” o de lo contrario lo clasificaremos como “normal”.
El reto en este caso es que “a simple vista”, es decir inspeccionando la señal ECG, no resulta fácil determinar si el sujeto es normal o anormal:
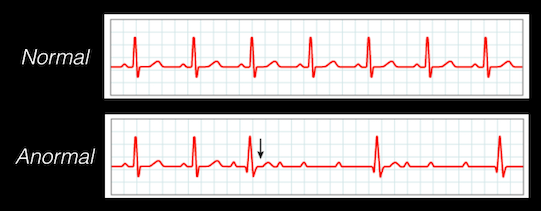
Adicionalmente, otro de los retos en este tipo de aplicaciones es que usualmente se cuenta con sets de datos desbalanceados, es decir que contienen más datos normales que anormales, lo que hace que resulte adecuado el uso de autoencoders para la solución de este problema.
El set de datos
Para entrenar nuestro autoencoder usaremos el set ECG5000 que contiene 7.600 datos de entrenamiento y 1.900 de prueba.
Cada uno de los datos ha sido pre-procesado siguiendo estos pasos:
- Extracción de un ciclo cardiaco
- Interpolación del ciclo extraído para garantizar que tendrá una longitud (número de muestras) fija.
En este último caso el set de datos fue pre-procesado de manera tal que cada una de las señales del set de datos contiene un total de 140 muestras, lo que permitirá diseñar nuestro autoencoder con una entrada de tamaño fijo.
Además, cada uno de los datos puede pertenecer a una de estas 5 categorías:
- Normal
- Anormal con contracción ventricular prematura
- Anormal con contracción supra-ventricular prematura
- Anormal latido ectópico
- Anormal pero patología desconocida
¿Por qué usar autoencoders en este caso?
El problema de este set de datos es que está desbalanceado, pues contiene 4427 datos normales y 3173 anormales.
Así que si usaramos un clasificador convencional (como por ejemplo un un clasificador con Redes Neuronales) tendríamos un sesgo: el modelo clasificaría mejor los datos “normales” que los “anormales”.
En lugar de esto usaremos un autoencoder, una arquitectura que es entrenada para aprender a reconstruir el dato de entrada, y que consta de estos elementos:
- Un encoder (o codificador) que permite obtener una representación compacta (es decir con menos datos) del dato de entrada
- El bottleneck (o cuello de botella, resultado de la compresión) que es como tal la representación compacta obtenida
- Y el decoder, que permite reconstruir el dato de entrada a partir de su representación compacta)
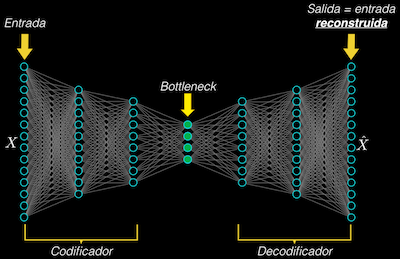
De esta forma, si entrenamos el autoencoder únicamente con los datos normales (la mayoría de los datos), aprenderá a reconstruirlos con una alta precisión, es decir con un error relativamente bajo.
Sin embargo, si posteriormente introducimos a este mismo autoencoder ya entrenado un dato anormal, el error de reconstrucción será relativamente alto (pues no ha sido entrenado para reconstruir este tipo de datos).
De esta manera tendremos un criterio de clasificación: si el error es “bajo” tendremos un dato “normal”, mientras que si el error es “alto” nuestro dato será “anormal”.
En lo que resta de este tutorial implementaremos y entrenaremos nuestro autoencoder y definiremos numéricamente los criterios para estos niveles de error “alto” y “bajo”.
Implementación del autoencoder
Lectura de los sets de entrenamiento y prueba
Haremos uso de la librería Pandas para la lectura de nuestros sets de entrenamiento y prueba, que se encuentran almacenados en formato csv (comma separated values):
ruta = '/gdrive/MyDrive/videos/2021-06-25/'
import pandas as pd
df_train = pd.read_csv(ruta + 'ECG5000_train.csv')
df_test = pd.read_csv(ruta + 'ECG5000_test.csv')
En este caso la variable ruta contiene, como su nombre lo indica, la ruta en donde se encuentran los archivos ECG5000_train.csv (set de entrenamiento) y ECG5000_test.csv (set de prueba).
Podemos verificar el tamaño de cada uno de estos sets:
print(df_train.shape)
print(df_test.shape)
obteniendo:
(7600, 141)
(1900, 141)
Es decir que el set de entrenamiento contiene 7.600 datos (filas), mientras que el segundo un total de 1.900.
Cada registro contendrá un total de 141 muestras (columnas):
- La primera columna nos indicará la categoría a la que pertenece el sujeto: 1 si el sujeto es “normal”, o un número entre 2 y 5 si el sujeto pertenece a cualquiera de las categorías “anormales” mencionadas anteriormente.
- Las 140 columnas restantes corresponden como tal a las muestras provenientes del ECG.
Sets de entrenamiento y validación
En primer lugar tomaremos únicamente los valores de cada registro, a partir de los Dataframes de Pandas creados en la sección anterior:
datos_train = df_train.values
datos_test = df_test.values
A continuación crearemos los arreglos que contendrán la categoría de cada dato, teniendo en cuenta que dicha categoría se encuentra en la primera columna (índice 0) de cada registro:
cat_train = datos_train[:,0]
cat_test = datos_test[:,0]
Ahora, teniendo en cuenta que tendremos un total de 5 categorías, crearemos la misma cantidad de arreglos con los registros correspondientes tanto al set de entrenamiento como al de prueba:
# Registros set de entrenamiento
x_train_1 = datos_train[cat_train==1,1:] # Normales (cat. 1)
x_train_2 = datos_train[cat_train==2,1:] # Anormales (cat. 2)
x_train_3 = datos_train[cat_train==3,1:] # Anormales (cat. 3)
x_train_4 = datos_train[cat_train==4,1:] # Anormales (cat. 4)
x_train_5 = datos_train[cat_train==5,1:] # Anormales (cat. 5)
# Registros set de prueba
x_test_1 = datos_test[cat_test==1,1:]
x_test_2 = datos_test[cat_test==2,1:]
x_test_3 = datos_test[cat_test==3,1:]
x_test_4 = datos_test[cat_test==4,1:]
x_test_5 = datos_test[cat_test==5,1:]
Pre-procesamiento (escalamiento)
Originalmente los rangos de amplitud en cada subset (x_train_1, x_train_2, …, x_test_5) no necesariamente serán los mismos, así que debemos garantizar que nuestros datos se encuentran dentro de la misma escala de amplitud para así facilitar el entrenamiento del autoencoder.
Este procedimiento se conoce como escalamiento y para ello usaremos como referencia el primer set de entrenamiento (x_train_1, datos “normales”), que se encuentra originalmente dentro de este rango:
print('Mínimo y máximo originales: {:.1f}, {:.1f}'.format(np.min(x_train_1), np.max(x_train_1)))
Mínimo y máximo originales: -7.1, 5.0
La idea es entonces tomar este rango como referencia para escalar todos los sets tanto de entrenamiento como de prueba al nuevo rango de 0 a 1.
Para lo anterior haremos uso de la librería Scikit-learn y en particular del módulo MinMaxScaler, que fácilmente nos permitirá hacer este pre-procesamiento.
Tengamos en cuenta que en el caso del set de entrenamiento basta con escalar tan sólo el set x_train_1, que contiene sólo los registros “normales”. Esto debido a que entrenaremos nuestro autoencoder sólo con este tipo de datos:
from sklearn.preprocessing import MinMaxScaler
min_max_scaler = MinMaxScaler()
# En primer lugar creamos una instancia y la entrenamos
# con el set de interés (x_train_1)
x_train_1_s = min_max_scaler.fit_transform(x_train_1)
# Y luego sí realizamos el escalamiento de todos los sets
x_test_1_s = min_max_scaler.transform(x_test_1)
x_test_2_s = min_max_scaler.transform(x_test_2)
x_test_3_s = min_max_scaler.transform(x_test_3)
x_test_4_s = min_max_scaler.transform(x_test_4)
x_test_5_s = min_max_scaler.transform(x_test_5)
Y podemos verificar por ejemplo que, tras este procedimiento, el set x_train_1_s estará en la escala deseada:
print('Mínimo y máximo normalización: {:.1f}, {:.1f}'.format(np.min(x_train_1_s), np.max(x_train_1_s)))
Mínimo y máximo escalamiento: 0.0, 1.0
Creación del Autoencoder en TensorFlow/Keras
Con los sets de entrenamiento y prueba ya listos podemos ahora crear nuestro modelo con ayuda de la librería TensorFlow que a su vez contiene la librería Keras para facilitar esta implementación.
En primer lugar importaremos las librerías requeridas:
from tensorflow.keras.models import Model
from tensorflow.keras.layers import Input, Dense
A continuación fijamos la semilla del generador aleatorio, lo que permitirá inicializar los coeficientes del autoencoder de forma aleatoria pero siempre en los mismos valores, con lo cual podremos repetir el entrenamiento una y otra vez obteniendo en todos los casos los mismos resultados:
np.random.seed(23)
A continuación creamos la capa de entrada al autoencoder, que tendrá exactamente el mismo tamaño de cada uno de los registros ECG (es decir 140 muestras):
dim_entrada = x_train_1_s.shape[1]
entrada = Input(shape=(dim_entrada,))
Ahora crearemos el encoder, que tendrá tres capas con 32, 16 y 8 neuronas respectivamente y con función de activación ReLU:
encoder = Dense(32, activation='relu')(entrada)
encoder = Dense(16, activation='relu')(encoder)
encoder = Dense(8, activation='relu')(encoder)
Este encoder se encarga de reducir la dimensionalidad del dato de entrada, pasando de 140 datos a un vector de 8 elementos, que corresponde precisamente al tamaño del bottleneck.
El siguiente elemento es el decoder, que realiza el proceso inverso del encoder: toma la representación compacta proveniente del bottleneck (es decir un vector de 8 elementos) y progresivamente incrementará su dimensionalidad hasta generar a la salida un vector de 140 elementos (el mismo tamaño del dato de entrada).
Esta implementación es muy similar a la del caso anterior y en este caso seguiremos usando el módulo Dense para crear el decodificador, que contendrá tres capas con 16, 32 y 140 neuronas respectivamente y también con función de activación ReLU (exceptuando la última capa que tendrá función de activación sigmoidal para que los valores de salida estén entre 0 y 1):
decoder = Dense(16, activation='relu')(encoder)
decoder = Dense(32, activation='relu')(decoder)
decoder = Dense(140, activation='sigmoid')(decoder)
Habiendo creado las etapas de codificación y decodificación, sólo nos resta interconectarlas para de esta forma tener nuestro autoencoder implementado:
autoencoder = Model(inputs=entrada, outputs=decoder)
Al escribir autoencoder.summary() podremos verificar que hemos construido correctamente el modelo y que este tendrá un poco más de 10.000 parámetros de entrenamiento:
Model: "model"
_________________________________________________________________
Layer (type) Output Shape Param #
=================================================================
input_1 (InputLayer) [(None, 140)] 0
_________________________________________________________________
dense (Dense) (None, 32) 4512
_________________________________________________________________
dense_1 (Dense) (None, 16) 528
_________________________________________________________________
dense_2 (Dense) (None, 8) 136
_________________________________________________________________
dense_3 (Dense) (None, 16) 144
_________________________________________________________________
dense_4 (Dense) (None, 32) 544
_________________________________________________________________
dense_5 (Dense) (None, 140) 4620
=================================================================
Total params: 10,484
Trainable params: 10,484
Non-trainable params: 0
Pérdida y Entrenamiento
Antes de realizar el entrenamiento debemos definir nuestra métrica de desempeño (es decir la pérdida).
Tanto a la entrada como a la salida del autoencoder tendremos secuencias de 140 elementos y lo que nos interesa es que el modelo aprenda a reconstruir correctamente los datos “normales”, mientras que en el caso de los “anormales” es de esperar que la reconstrucción no sea tan buena.
Teniendo en cuenta lo anterior lo que nos interesa es una métrica que permita comparar el vector de salida con el vector de entrada, para lo cual haremos uso del error absoluto medio (Mean Absolute Error o MAE), que se define como:
$MAE = \frac{\sum_{i=1}^{140} |x_i - \hat{x_i}|}{140}$
donde:
- $x_i$ es cada uno de los 140 datos en el vector de entrada (dato original)
- $\hat{x_i}$ es cada uno de los 140 datos en el vector de salida (dato reconstruido)
En esencia este MAE promedia los valores absolutos de las diferencias elemento-a-elemento entre la entrada y la salida del autoencoder. Así, si el dato reconstruido se asemeja bastante al dato original, es de esperar que este MAE sea cercano a cero.
Lo que resta ahora es usar el método compile para especificar precisamente la pérdida (loss='mae'), y además el tipo de optimizador a usar durante el entrenamiento (que en este caso será Adam: optimizer='adam'). Todo esto lo podemos hacer con tan solo una línea de código:
autoencoder.compile(optimizer='adam', loss='mae')
Finalmente, podemos realizar el entrenamiento del autoencoder usando el método fit. En este caso usaremos un total de 20 iteraciones (epochs=20) y un tamaño de lote de 512 (batch_size=512). De nuevo, ¡y esto es lo maravilloso de Keras!, todo esto requiere tan sólo una línea de código:
autoencoder.compile(optimizer='adam', loss='mae')
historia = autoencoder.fit(x_train_1_s, x_train_1_s,
epochs=20,
batch_size=512,
validation_data=(x_test_1_s, x_test_1_s),
shuffle=True)
Observemos que hemos especificado que tanto el dato de entrada como de salida serán los datos de entrenamiento correspondientes a los sujetos normales (x_train_1_s, x_train_1_s) y que en cada iteración usaremos los datos de prueba (también correspondientes a sujetos normales) para realizar la validación (validation_data=(x_test_1_s, x_test_1_s)).
Una vez realizado el entrenamiento podemos verificar si nuestro modelo posee algún tipo de underfitting u overfitting o si por el contrario ha sido entrenado correctamente.
Para lo anterior podemos usar la información almacenada en la variable historia para generar una gráfica de la pérdida (MAE, eje vertical) vs. el número de iteraciones (eje horizontal), tanto para los datos de entrenamiento como para los de validación:
plt.plot(historia.history["loss"], label="Pérdida set entrenamiento")
plt.plot(historia.history["val_loss"], label="Pérdida set prueba")
plt.legend();
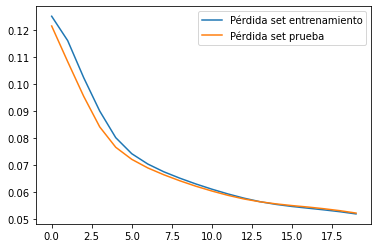
A partir de esta gráfica podemos concluir que no existe ni overfitting ni underfitting ¡pues las dos curvas prácticamente coinciden en la iteración 20!
Clasificación
Con el autoencoder ya entrenado podemos ponerlo a prueba para la detección de anomalías cardiacas.
Reconstrucción: análisis cualitativo
En primer lugar haremos un análisis cualitativo: reconstruiremos un dato “normal” (para el cual fue entrenado el autoencoder) y uno “anormal” y dibujaremos el error en la reconstrucción para cada caso.
Para lo anterior introducimos al modelo el set de prueba x_test_1_s (que contiene solo datos normales) y el set x_test_5_s (que contiene únicamente datos anormales):
rec_normal = autoencoder(x_test_1_s).numpy()
rec_anormal = autoencoder(x_test_5_s).numpy()
y ahora sí generamos las dos gráficas de reconstrucción para el dato 5, usando la librería Matplotlib:
dato = 5
plt.figure(figsize=(15,5))
plt.subplot(1,2,1)
plt.plot(x_test_1_s[dato],'b')
plt.plot(rec_normal[dato],'r')
plt.fill_between(np.arange(140), rec_normal[dato], x_test_1_s[dato], color='lightcoral')
plt.legend(labels=["Original normal", "Reconstruction", "Error"])
plt.subplot(1,2,2)
plt.plot(x_test_5_s[dato],'b')
plt.plot(rec_anormal[dato],'r')
plt.fill_between(np.arange(140), rec_anormal[dato], x_test_5_s[dato], color='lightcoral')
plt.legend(labels=["Original anormal", "Reconstruction", "Error"])
donde en la anterior porción de código hemos usado la función fill_between que automáticamente calcula y dibuja con un color la diferencia entre los datos original y reconstruido.
El resultado de estas reconstrucciones es el siguiente:
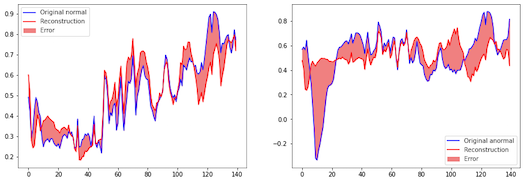
Podemos observar que el error en la reconstrucción (porción de color rojo en cada gráfica) es menor en el dato “normal” (izquierda) en comparación con el dato “anormal” (derecha). Esto nos permite confirmar, de forma cualitativa, que el autoencoder al parecer funciona correctamente pues genera mejores reconstrucciones para los datos con los cuales fue entrenado (es decir los datos “normales”).
Detección automática de anomalías cardiacas
Muy bien, habiendo verificado de manera subjetiva el desempeño del autoencoder ahora nos enfocaremos en la detección automática de anomalías en las señales ECG.
La lógica de esta etapa de clasificación es muy sencilla:
- Introducimos al autoencoder un dato que queramos clasificar, obtenemos la reconstrucción y calculamos el MAE (el error en la reconstrucción).
- Comparamos este MAE con un umbral pre-establecido: si el error es inferior a este umbral, clasificamos el dato como “normal”. Por el contrario, si el error es mayor o igual al umbral lo clasificaremos como “anormal”.
Así que el éxito de este clasificador radicará en la correcta selección del umbral.
Para escoger este umbral podemos usar el siguiente enfoque:
- Generamos las reconstrucciones de cada uno de los datos en el set de prueba (
x_test_1_sax_test_5_s) - Calculamos el MAE entre cada reconstrucción y el correspondiente dato original
- Si las distribuciones de error de los datos “anormales” están lo suficientemente separadas de las correspondientes a los “normales”, podemos simplemente definir el umbral como:
$umbral = \overline{MAE_1} + \sigma_{MAE_1}$
donde $\overline{MAE_1}$ es simplemente el promedio de los MAEs para los datos “normales” y $\sigma_{MAE_1}$ es su desviación estándar.
Con lo anterior estamos asumiendo que los errores tendrán una distribución normal y que aquellos errores que se alejen una desviación estándar de la media de los sujetos “normales” serán entonces clasificados como anormales.
Veamos entonces cómo calcular este umbral en Python según el procedimiento que acabamos de describir. En primer lugar obtengamos las reconstrucciones para cada subset en el set de prueba:
rec_1 = autoencoder.predict(x_test_1_s)
rec_2 = autoencoder.predict(x_test_2_s)
rec_3 = autoencoder.predict(x_test_3_s)
rec_4 = autoencoder.predict(x_test_4_s)
rec_5 = autoencoder.predict(x_test_5_s)
A continuación calculemos los errores (MAEs) obtenidos en cada reconstrucción (usando el módulo losses.mae de Tensorflow/Keras):
import tensorflow as tf
loss_1 = tf.keras.losses.mae(rec_1, x_test_1_s)
loss_2 = tf.keras.losses.mae(rec_2, x_test_2_s)
loss_3 = tf.keras.losses.mae(rec_3, x_test_3_s)
loss_4 = tf.keras.losses.mae(rec_4, x_test_4_s)
loss_5 = tf.keras.losses.mae(rec_5, x_test_5_s)
Ahora generemos la gráfica de las diferentes distribuciones de error con ayuda de Matplotlib:
plt.figure(figsize=(15,8))
plt.hist(loss_1[None,:], bins=100, alpha=0.75, label='normales (1)')
plt.hist(loss_2[None,:], bins=100, alpha=0.75, color='#ff521b', label='anormales (2)')
plt.hist(loss_3[None,:], bins=100, alpha=0.75, color='#020122', label='anormales (3)')
plt.hist(loss_4[None,:], bins=100, alpha=0.75, color='#eefc57', label='anormales (4)')
plt.hist(loss_5[None,:], bins=100, alpha=0.75, color='r', label='anormales (5)')
plt.xlabel('Pérdidas (MAE)')
plt.ylabel('Nro. ejemplos')
plt.legend(loc='upper right')
plt.vlines(0.08,0,70,'k')
Obteniendo este resultado:
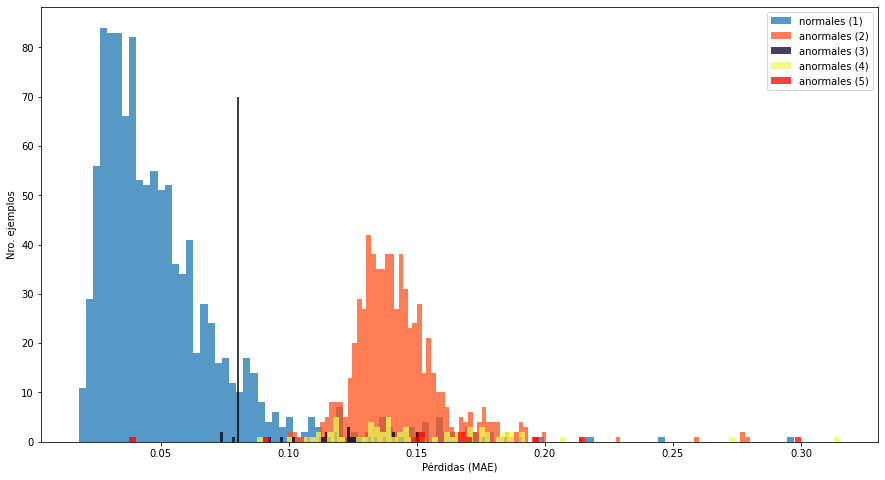
Podemos observar que la distribución correspondiente a los “normales” (la de mayor tamaño, en color azul a la izquierda) está relativamente separada de las agrupaciones restantes (de menor tamaño y correspondientes a los anormales). Así que nuestra hipótesis de usar un umbral para separar “normales” de “anormales” parece ser la adecuada.
En la gráfica anterior hemos dibujado un umbral ideal (línea vertical de color negro, ubicada en 0.08) que permitiría generar esta separación. Tengamos en cuenta que en todo caso no existirá una separación ideal, pues existe cierto solapamiento entre la distribución de “normales” y algunas de las correspondientes a los “anormales” (aunque esto lo analizaremos al final de este artículo).
Lo que nos resta entonces es calcular automáticamente el umbral usando la media y la desviación estándar de la distribución de errores para los “normales”:
umbral = np.mean(loss_1) + np.std(loss_1)
print("Umbral: ", umbral)
Obteniendo el siguiente resultado:
Umbral: 0.0828392467372577
Que coincide precisamente con el umbral mostrado en la gráfica anterior. Así que para el proceso de clasificación usaremos un umbral MAE = 0.08.
Para realizar la clasificación de un dato implementaremos una pequeña función (predecir) que calculará la reconstrucción y el error correspondiente, para luego compararlo con el umbral anterior y definir si el dato es “normal” o “anormal”:
def predecir(modelo, datos, umbral):
reconstrucciones = modelo(datos)
perdida = tf.keras.losses.mae(reconstrucciones, datos)
return tf.math.less(perdida, umbral)
donde hemos usado tf.math.less(perdida, umbral) para comparar el MAE (perdida) con el umbral establecido anteriormente (umbral): si perdida < umbral la categoría será “normal”, de lo contrario será clasificado como “anormal”.
Desempeño: sensitividad y especificidad
Bien, para finalizar este tutorial lo único que nos resta es evaluar el desempeño de este clasificador.
Teniendo en cuenta que se trata de una aplicación médica, debemos hacer esta evaluación a la luz de los criterios comúnmente usados para el caso de herramientas de diagnóstico.
Estos criterios son la sensitividad y la especificidad. La sensitividad mide la proporción de “anormales” que fueron detectados correctamente como “anormales”, mientras que la especificidad mide la proporción de normales que fueron detectados correctamente como normales.
Para calcular estas dos métricas debemos determinar previamente la cantidad de:
- Verdaderos positivos (TP): anormales que han sido correctamente clasificados como anormales
- Falsos negativos (FN): anormales que han sido clasificados erróneamente como normales
- Verdaderos negativos (TN): normales que han sido correctamente clasificados como normales
- Falsos positivos (FP): normales que han sido clasificados erróneamente como anormales
Teniendo estos parámetros podemos fácilmente calcular la sensitividad y la especificidad de la siguiente forma:
$sensitividad = \frac{TP}{TP+FN}\cdot 100%$
$especificidad = \frac{TN}{TN+FP}\cdot 100%$
Así, una sensitividad ideal del 100% indicará que el clasificador es capaz de detectar a todos los pacientes enfermos, mientras que una especificidad del 100% indicará que todos los pacientes sanos serán clasificados correctamente.
En la práctica es casi imposible alcanzar estos niveles ideales en ambos casos, y para nuestro problema en particular no será la excepción (pues recordemos que no existe una perfecta separación entre las distribuciones de error para “normales” y “anormales”).
Vamos de todos modos qué tan cerca estamos de estos desempeños ideales. En primer lugar, y con ayuda de Numpy, podemos crear dos sencillas funciones para calcular la sensitividad y especificidad a partir de las predicciones generadas con la función predecir que vimos anteriormente:
def calcular_sensitividad(prediccion, titulo):
TP = np.count_nonzero(~prediccion)
FN = np.count_nonzero(prediccion)
sen = 100*(TP/(TP+FN))
print(titulo + ': {:.1f}%'.format(sen))
def calcular_especificidad(prediccion, titulo):
TN = np.count_nonzero(prediccion)
FP = np.count_nonzero(~prediccion)
esp = 100*(TN/(TN+FP))
print(titulo + ': {:.1f}%'.format(esp))
Y a continuación generemos las predicciones para cada subset en el set de prueba usando el umbral de 0.08 definido anteriormente:
pred_1 = predecir(autoencoder, x_test_1_s, umbral)
pred_2 = predecir(autoencoder, x_test_2_s, umbral)
pred_3 = predecir(autoencoder, x_test_3_s, umbral)
pred_4 = predecir(autoencoder, x_test_4_s, umbral)
pred_5 = predecir(autoencoder, x_test_5_s, umbral)
Ahora obtenemos la especificidad, que por definición se calcula únicamente sobre los datos “normales”:
calcular_especificidad(pred_1,'Especificidad (cat. 1, normales)')
Obteniendo un valor de 89.3% que es bastante cercano a nuestro ideal del 100%. Este resultado nos indica que nuestro autoencoder clasificará a los sujetos “normales” realmente como “normales” en aproximadamente el 89% de los casos.
Veamos lo que ocurre con la sensitividad que se calcula únicamente sobre los datos “anormales”. En este caso haremos cuatro cálculos, uno para cada subcategoría de anormales:
calcular_sensitividad(pred_2,'Sensitividad (cat. 2, anormales)')
calcular_sensitividad(pred_3,'Sensitividad (cat. 3, anormales)')
calcular_sensitividad(pred_4,'Sensitividad (cat. 4, anormales)')
calcular_sensitividad(pred_3,'Sensitividad (cat. 5, anormales)')
obteniendo estos resultados:
Sensitividad (cat. 2, anormales): 100.0%
Sensitividad (cat. 3, anormales): 90.9%
Sensitividad (cat. 4, anormales): 100.0%
Sensitividad (cat. 5, anormales): 90.9%
Vemos que el desempeño es incluso mejor que el obtenido con la especificidad. En particular las categorías 2 y 4 son clasificadas correctamente como anormales en el 100% de los casos, mientras que en las categorías 3 y 5 este porcentaje se reduce casi al 91% pero aún sigue siendo bastante bueno.
Las sensitividades del 100% se pueden explicar por el hecho de que las distribuciones de las categorías 2 y 5 se encuentran perfectamente separadas de la categoría “normal” y por este motivo el umbral de 0.08 permite alcanzar una separación ideal.
¡Y listo, ya hemos logrado implementar y poner a prueba este sistema de detección de anomalías cardiacas!
Conclusión
Bien, en este tutorial vimos cómo implementar y poner a prueba un autoencoder capaz de detectar anomalías cardiacas a partir de señales ECG.
Al ponerlo a prueba pudimos verificar que tiene un desempeño bastante bueno, logrando clasificar correctamente a los sujetos “normales” el 89% de las veces, mientras que en el caso de los anormales se alcanzan porcentajes de entre el 90% e incluso el 100%.
Si tienes alguna duda de este artículo o tienes alguna sugerencia no dudes en contactarme diligenciando el siguiente formulario:
Código fuente
En este enlace de Github podrás descargar el código fuente y el set de datos de este tutorial.